Le logiciel MassXpert (actuellement MassXpert3) est un programme de modélisation de la chimie des polymères linéaires qui vise à simuler des données de spectrométrie de masse pour des séquences (bio) polymériques : une fois qu’une définition de la chimie des polymères a été modélisée, une séquence polymérique peut être saisie dans l’éditeur et un grand nombre de réactions de chimie des polymères peut être simulé avec production des données de spectrométrie de masse théoriques correspondantes.
MassXpert comporte les modules suivants
XpertDef
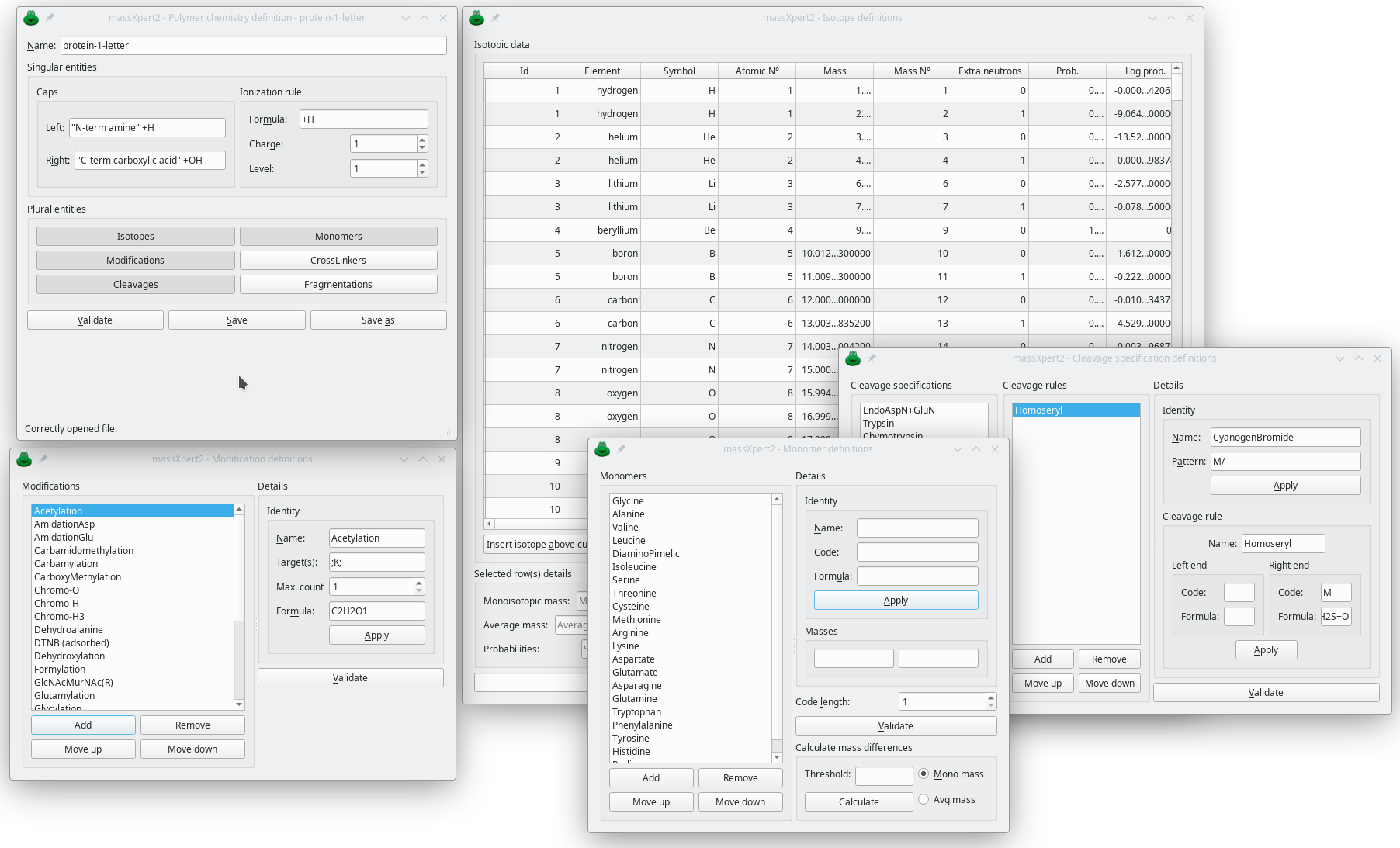
Avec ce module, l’utilisateur définit de toutes nouvelles chimies de polymères. Au minimum, une chimie de polymère comprend les informations chimiques suivantes :
-
Une table de données isotopiques (isotopes nécessaires pour décrire les éléments chimiques)
-
Une liste de monomères définis par un code (n’importe quel nombre de lettres), un nom et une formule. La formule définit le résidu, pas le monomère.
-
Une liste de modifications (bio) chimiques définies par un nom, une formule chimique nette (la différence atomique nette entre les états non modifié et modifié), une liste de cibles de monomères qui pourraient être modifiées avec cette modification, et enfin le nombre maximum de telles modifications pouvant se produire sur une seule cible de monomère (pour la méthylation, cela pourrait être 3, par exemple, dans les histones ; ou pour la phosphorylation, cela pourrait être 1, pour les résidus de thréonine, sérine, tyrosine).
-
Une liste d’agents de clivage (chimiques ou biochimiques), comme les enzymes protéolytiques ou les enzymes endonucléolytiques, mais aussi comme le bromure de cyanogène. La flexibilité de la grammaire utilisée pour définir les modifications permet la définition de modifications de monomères se produisant lors de l’utilisation de réactifs chimiques comme le bromure de cyanogène, par exemple, où le résidu méthionyl est converti en un résidu homoséryl.
-
Une liste de schémas de fragmentation en phase gazeuse pour la simulation détaillée de la fragmentation d’ions précurseurs de la définition de polymère correspondante. La grammaire utilisée pour la définition des schémas de fragmentation en phase gazeuse est si flexible qu’elle permet la description des mécanismes de fragmentation les plus complexes dans le monde des saccharides.
-
Les extrémités de séquence de polymères. Dans le monde des peptides/protéines, le polymère est une chaîne de résidus de monomères qui, pour correspondre à une séquence de polymère finie, doit être coiffée à la fois d’un proton à l’extrémité N-terminale et d’un groupe hydroxyle à l’extrémité C-terminale.
-
La règle d’ionisation par défaut à utiliser lors du calcul de la composition élémentaire et des masses du polymère ou de l’oligomère (protéine ou peptide, par exemple). Une règle d’ionisation est définie en spécifiant la modification chimique à laquelle l’analyte est soumis lors de l’ionisation, la charge apportée par cette réaction d’ionisation et enfin le nombre de fois que la réaction d’ionisation doit être effectuée sur l’analyte. Pour la spectrométrie de masse MALDI des protéines, cela serait typiquement [+H, 1, 1], ce qui se lit comme “La protonation apporte un proton à l’analyte, apportant ainsi une charge de un une seule fois”.
XpertCalc
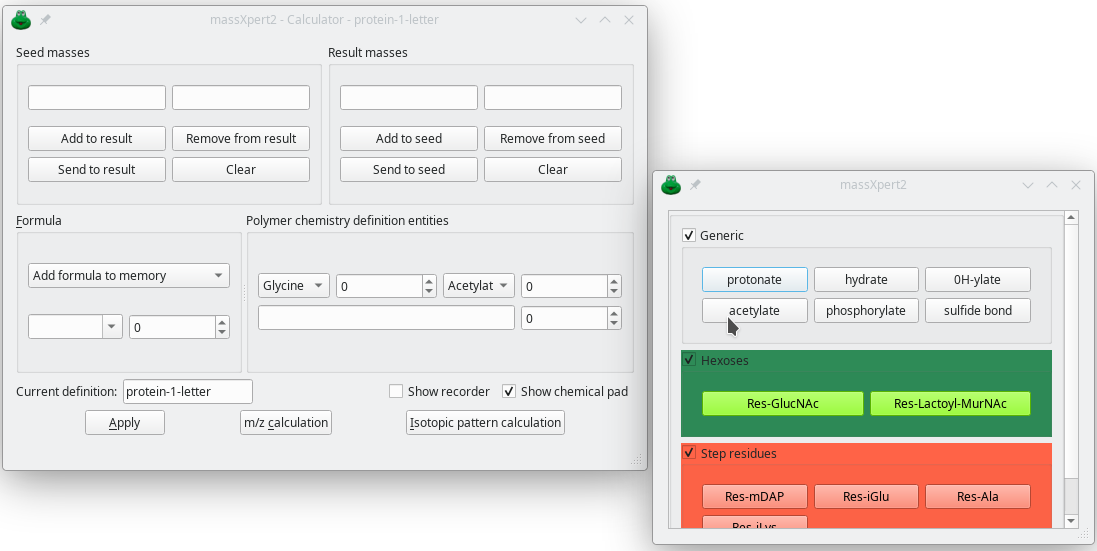
Avec ce module, vous obtenez une calculatrice de bureau qui comprend vos définitions de chimie des polymères telles que définies dans XpertDef. La calculatrice permet tout type de réaction chimique et est infiniment programmable. Un pad chimique est disponible dans lequel les réactions chimiques précédemment définies deviennent disponibles sous forme de boutons, exactement comme avec une calculatrice de bureau. Toute calcul est enregistré dans un journal qui est exportable pour être intégré dans le cahier de laboratoire.
XpertEdit
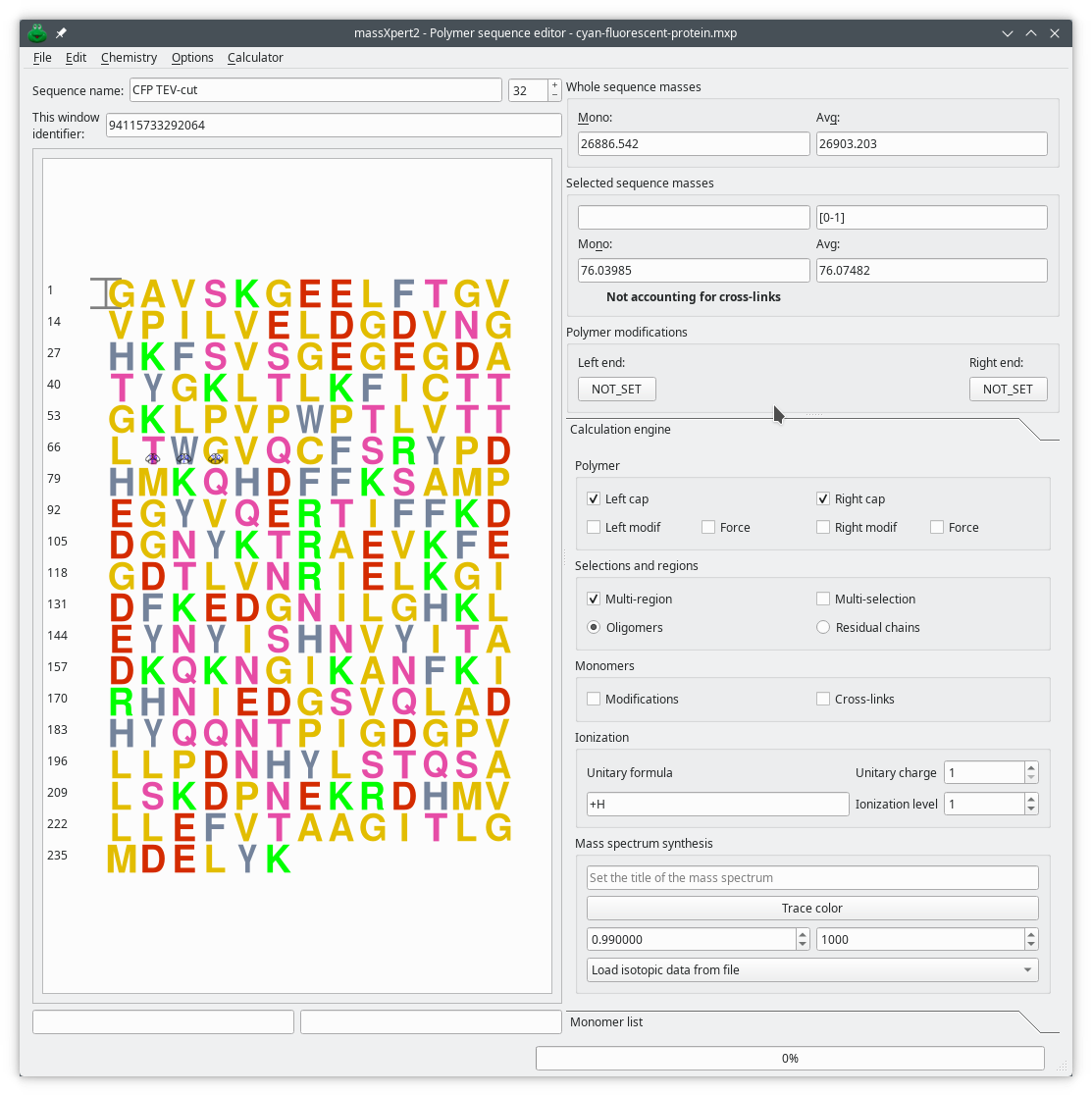
Avec ce module, vous disposez d’un éditeur de séquences polymériques sophistiqué et d’un centre chimique où une énorme quantité de simulations peut être effectuée. Tout ce qui concerne la masse est pratiquement réalisable dans XpertEdit. L’éditeur de séquences polymériques est configurable dans la mesure où la représentation graphique des résidus de monomères dans la séquence est définissable à l’aide d’un programme de dessin qui produit des fichiers SVG (comme Inkscape](https://www.inkscape.org), par exemple).
Simulation de massif isotopique
La simulation de massifs isotopiques à partir d’une formule, classiquement assez complexe, est rendu facile par l’interface disponible dans MassXpert. Il suffit d’insérer la formule de la molécule analysée (en y ajoutant le nombre d’agents d’ionisation —typiquement des protons pour l’analyse de protéines):
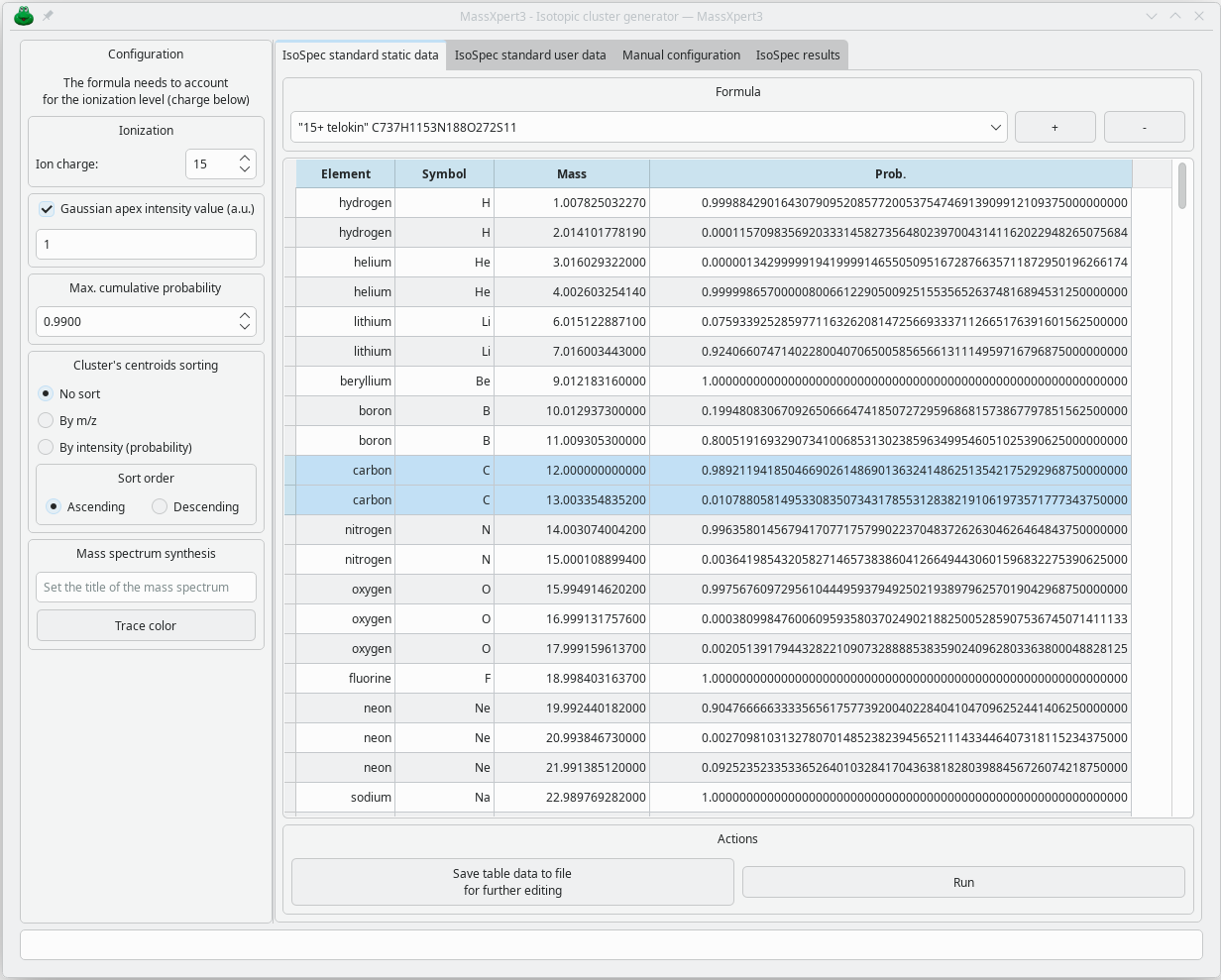
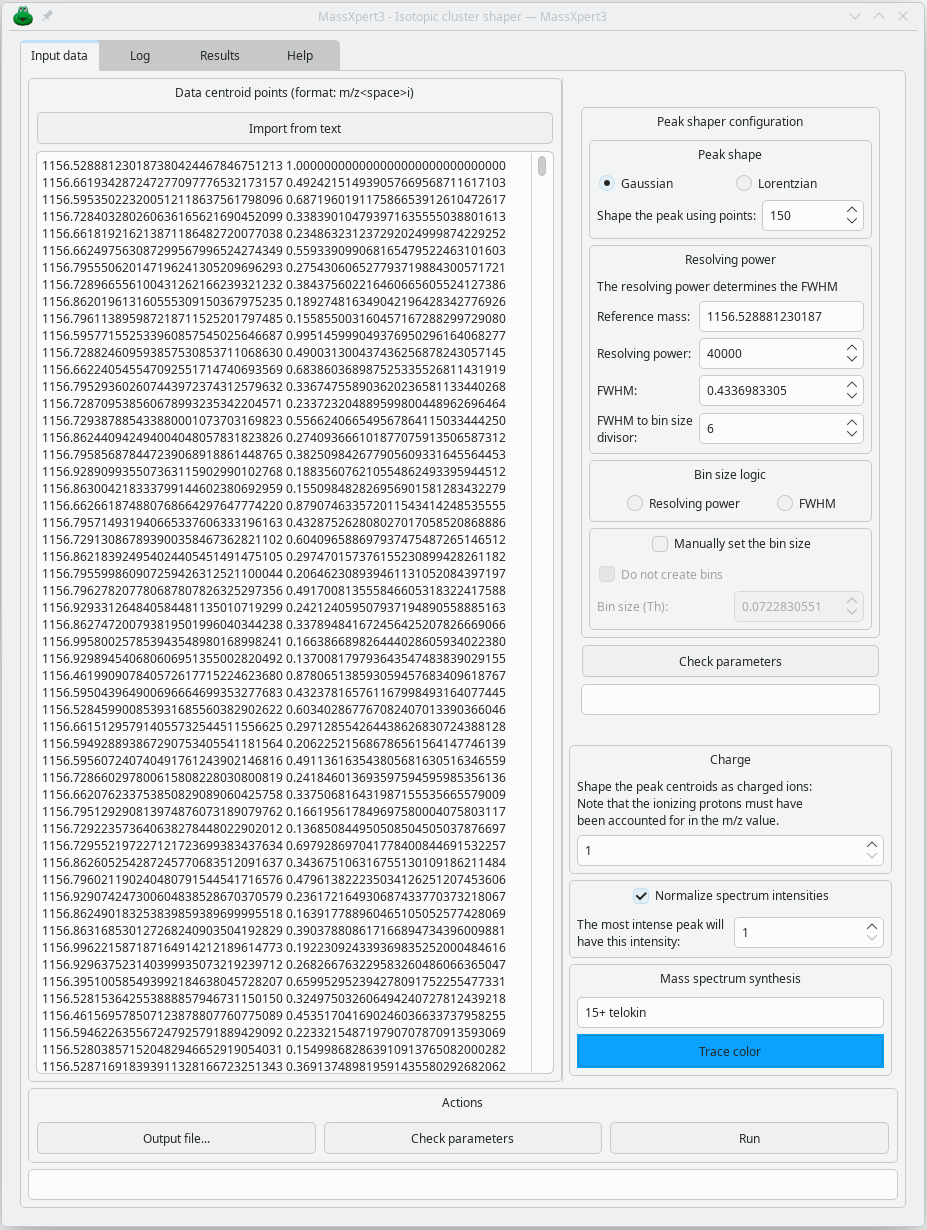
Une fois que ce paramétrage est fait, le massif isotopique se présente comme un histogramme dont les valeurs en x sont les m/z et les valeurs en y les probabilités. Mais ces valeurs ne sont que des centroïdes et ne donnent pas une idée de l’aspect du massif isotopique avec son profil en relation avec le pouvoir résolutif de l’appareil. Pour obtenir le bon profil, il faut utiliser la fonctionnalité illustrée ci-dessous:
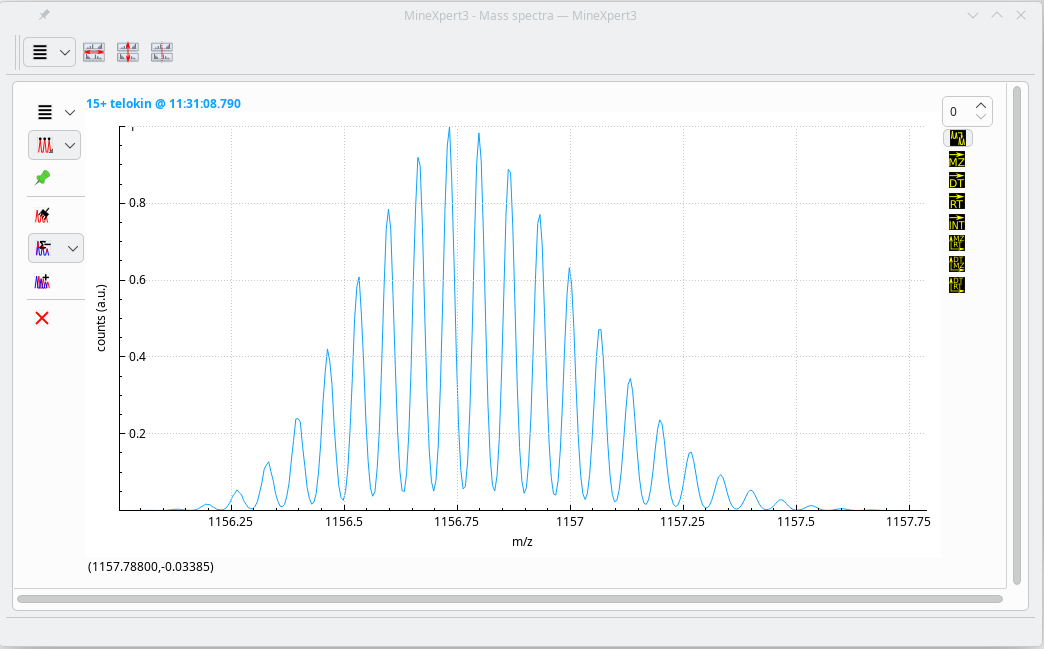
Une fois que le spectre est généré comme attendu par un spectromètre de masse d’un pouvoir résolutif donné, il est affiché dans le programme MineXpert3:
Clivage et simulation de spectre
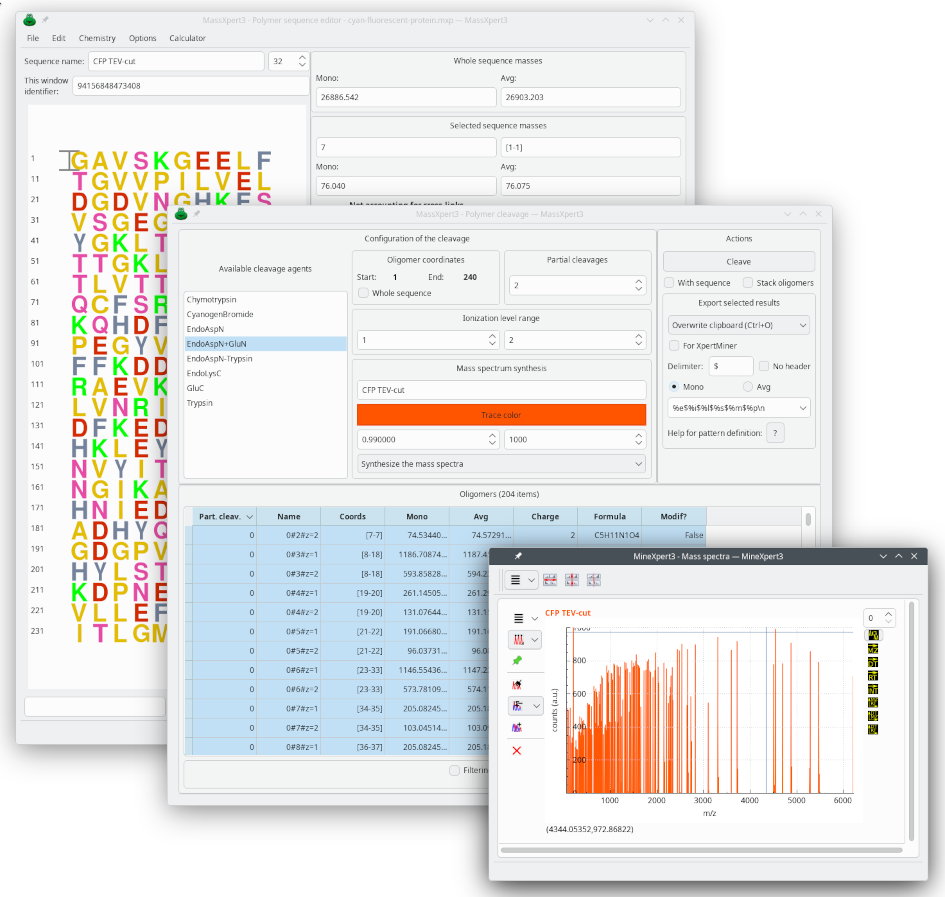
Lorsque la séquence de polymère est prête dans l’éditeur de séquence, il est possible, parmi de nombreuses autres possibilités, de l’hydrolyser avec l’enzyme (ou le cocktail d’enzymes) de son choix. Ensuite les oligomères peuvent être utilisés pour simuler un spectre de masse de l’hydrolysat en fonction du pouvoir résolutif de l’instrument. Ce ci est illustré dans la figure ci-dessous, où le spectre de masse synthétique a été automatiquement affiché dans MineXpert:
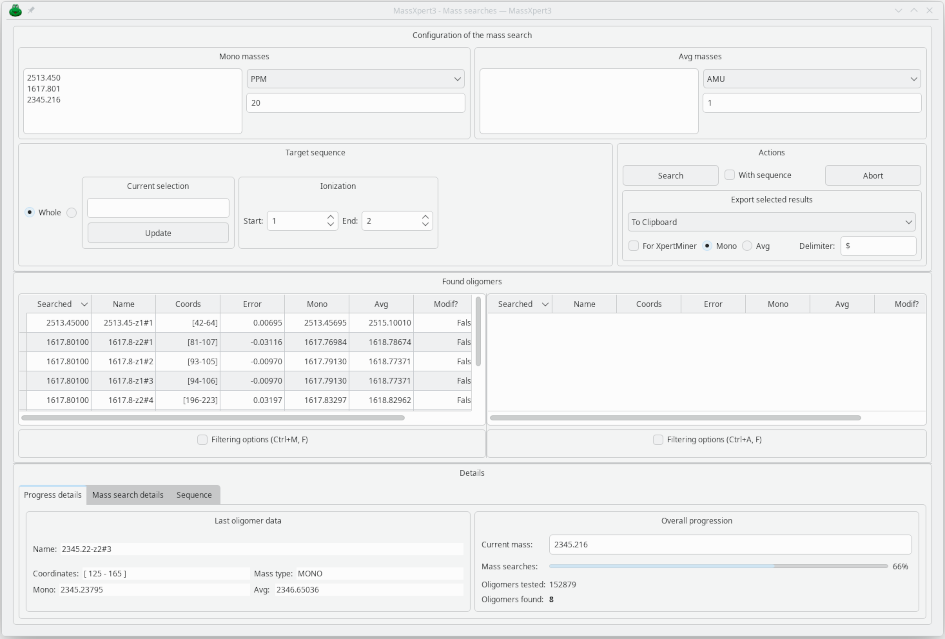
Recherches de masses arbitraires
Il est possible d’effectuer des recherches de masse (de m/z) sur une séquence donnée. Typiquement, cela est utile quand certains pics de masse ne peuvent pas être expliqués par une digestion enzymatique spécifique:
JavaScript
Avec le module JavaScript, vous disposez d’un environnement de script de données dans lequel la plupart des fonctionnalités de MassXpert sont accessibles depuis l’environnement ECMAScript.
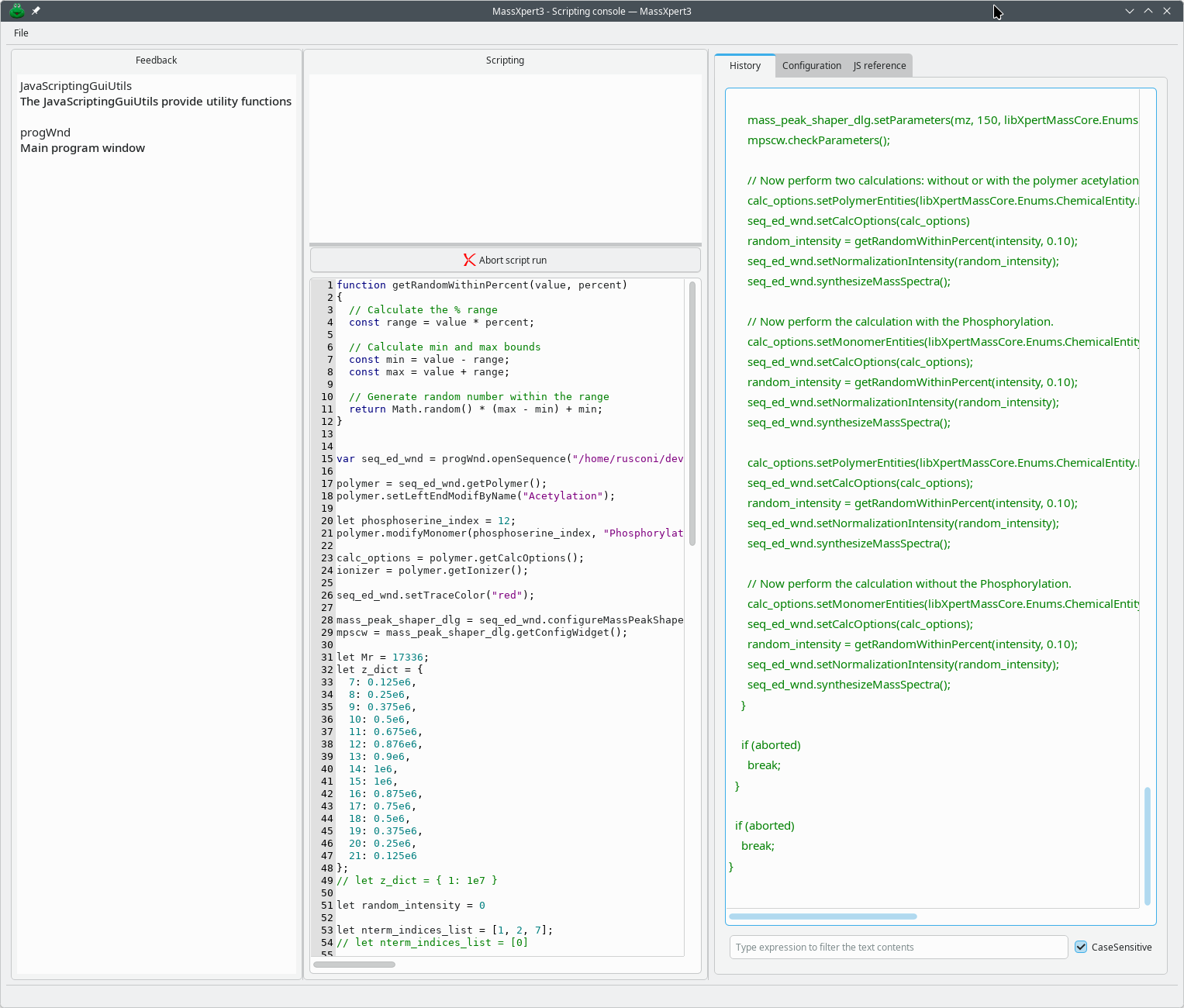
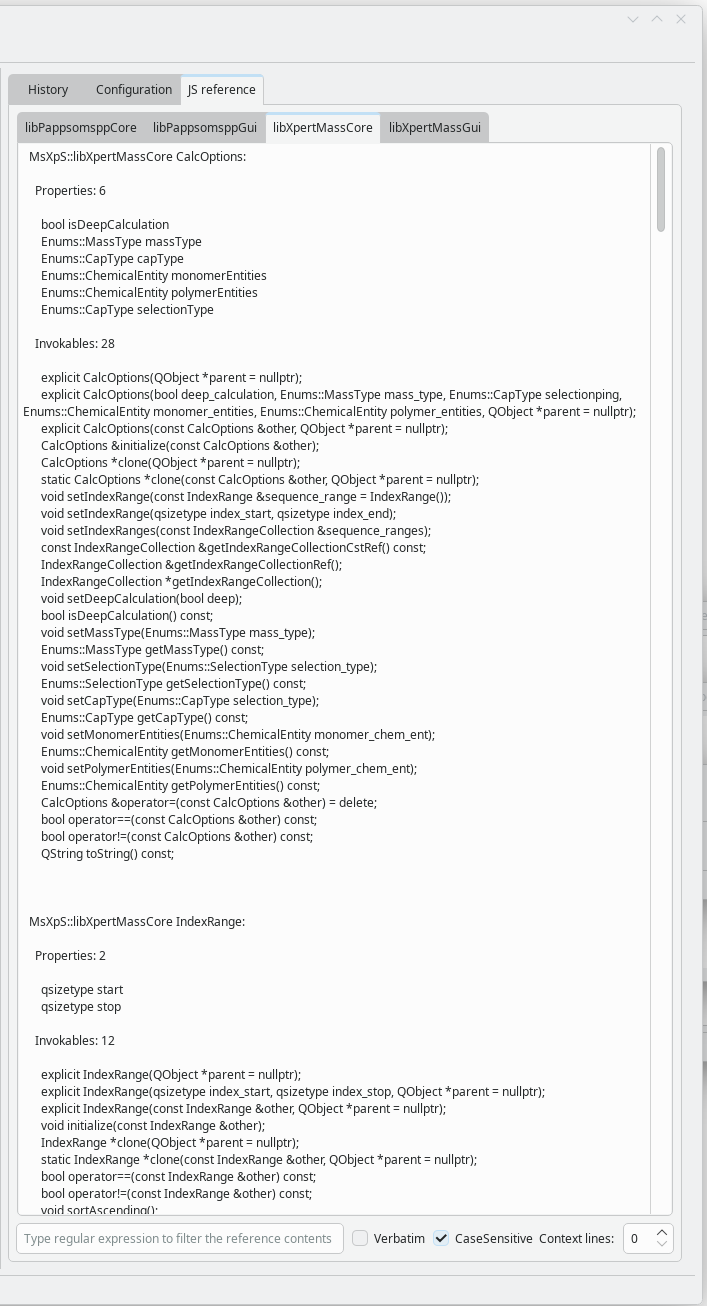
La documentation JavaScript est extraite de balises dédiées dans le code source et est donc fiable. Elle est affichée pour chaque composant (en particulier les bibliothèques) dans l’onglet Référence JS, comme illustré ci-dessous :